两篇胃癌lncRNA signature文章解读之一(IF=7.7)
看教程不够直观,那就看视频吧! >>点击加载视频
这是一篇2016年的文章,文章从lncRNA的角度筛选lncRNA signature,该文章发表在Molecular Cancer上,影响因子如下:

下面我们一起来解析一下这篇文章
文章标题如:

看signature大体思路基本上都是
1、整理数据
2、初步筛选基因
3、进一步降低维度到几个或者十几二十个
4、建立预后模型
5、外部数据验证
基于以上套路,我们开始看这篇文章
从方法部分揣测思路:
 从中可以看出本文使用lncRNA表达谱数据集为(GSE62254、GSE15459)两套数据集,筛选了24个lncRNA的panel,用来对病人进行分类,使用无病生存期(DFS)这个指标来衡量分类的病人的生存期长短。
从中可以看出本文使用lncRNA表达谱数据集为(GSE62254、GSE15459)两套数据集,筛选了24个lncRNA的panel,用来对病人进行分类,使用无病生存期(DFS)这个指标来衡量分类的病人的生存期长短。
基本上大体思路和结论便有了:两套数据集一套做验证,一套做训练,从中提取lncRNA的表达谱,来筛选预后相关的lncRNA,构建预后模型。
再看看结果:

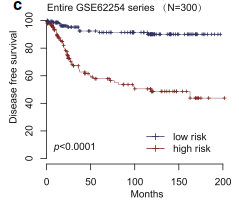
基于这24个lncRNA能够对样本进行高低风险分类,高风险和低风险样本的DFS具有显著的差异(p<0.0001),在验证数据集中得到了确认;然而这个分类模型与LNR和化疗没有关系,进一步的做GSEA功能富集分析发现高风险样本主要富集在癌症复发和转移相关的通路中(在功能上与分类结果吻合)
作者的具体做法是怎么做的呢?我们从材料与方法中探索一下
1、数据下载:
从GEO数据库(http://www.ncbi.nlm.nih.gov/geo/)中下载GSE62254、GSE15459两套数据集的原始数据CEL(这两套数据集都带有预后信息),从GSE62254数据集(N=300)中随机的分成两组一组作为训练集(N=180),一组作为测试集(N=120);这里随机分组比例为3:2,然后使用GSE15459数据作为外部数据集。
2、数据的处理
首先去除掉没有预后信息的样本,再进一步从原始数据CEL开始处理,统一使用RMA标准化来对数据进行标准化,然后从中提取lncRNA,提取方法参考了之前的一篇文章:Long non-coding RNA expression profiles predict clinical phenotypes in glioma,首先根据NetAffx的注释文件将探针ID映射到Ensembl gene ID 然后根据基因的属性文件来识别non-coding protein genes,去除pseudogenes 、microRNAs, rRNAs and other short RNAs such as snoRNAs, snRNAs and tRNAs;最终得到了2448个lncRNA的表达谱。
3、单因素生存分析
首先对训练集中每个lncRNA的表达谱使用BRB-Array工具进行单因素生存分析,选择阈值为p<0.01,共得到了63个lncRNA
4、进一步降维
63个lncRNA用了做预后模型显然太多,不利于临床应用,所以作者进一步的对这63个基因进行降维缩小模型中lncRNA的个数,使用随机网络森林变量搜索算法(RSF-VH)来进行进一步的降维,这个算法该是一种降维方法,用于度量生存树中变量的预测能力,在这种情况下进行有效的变量选择,最终得到了24个预测能力很好的lncRNA(这一步可以使用R包randomForestSRC来实现),然后通过查阅文献分析了这24个lncRNA有没有被人报道过与肿瘤的关系,如表中第七列。
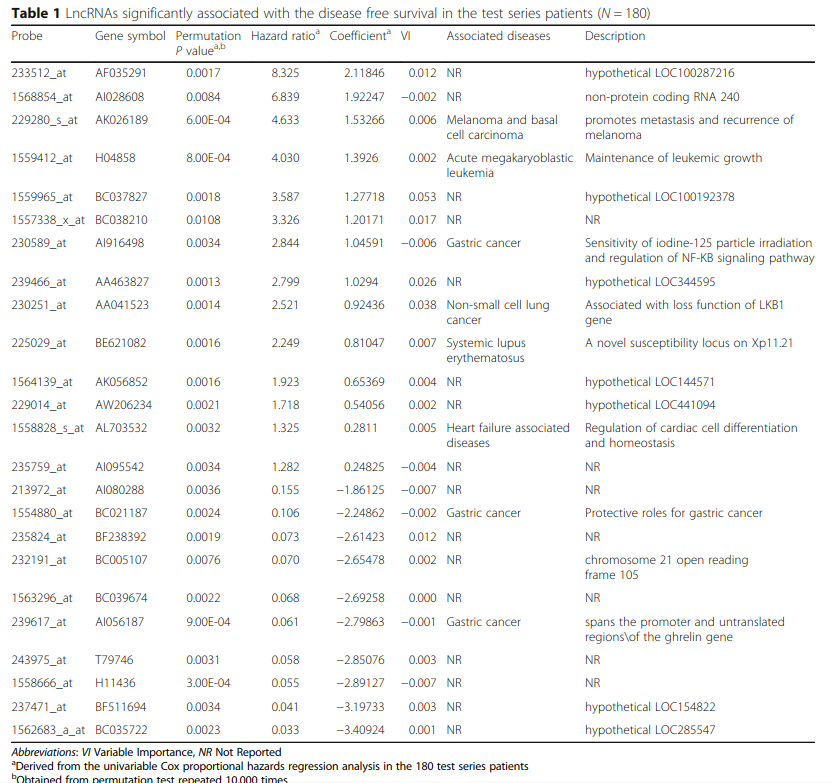
5、建立预后模型
根据RSF-VH算法得到了24个lncRNA,也得到了这24个lncRNA对应的模型的系数,如表格Table1中的第五列,根据这个系数来建立预后模型

6、根据模型计算每个样本的风险得分
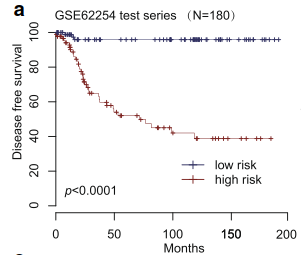
模型公式已经得到了,这个时候我们只需要将每个样本中对应的那24个lncRNA的表达水平提取出来,然后带入这个公式就可以得到这个样本的风险得分了,这个时候我们共有180个样本就得到了180个样本的风险得分,我们根据这得分的高低对样本进行平均分类,分成得分高的一组90个,得分低的一组90个,然后再进一步分析两组的预后差异,发现他们具有显著的预后差异如图:
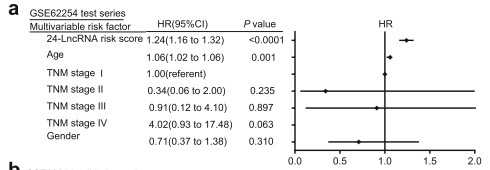
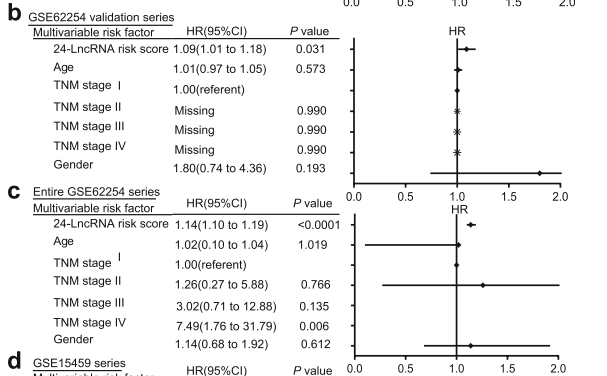
 进一步的我们比较了这些风险得分的预后价值与临床其他特征(TNM)的预后价值,如图,可以看出这个风险模型的p值极显著,而其他几个临床特征p值很大,这说明这个模型的预后价值比TNM Stage好。
进一步的我们比较了这些风险得分的预后价值与临床其他特征(TNM)的预后价值,如图,可以看出这个风险模型的p值极显著,而其他几个临床特征p值很大,这说明这个模型的预后价值比TNM Stage好。
7、测试数据集验证
我们用测试数据集中的这24个lncRNA的表达谱来计算每个样本的风险得分,然后使用中位数来看一下能不能对样本进行高低风险分析如图,发现也能够将样本分开。

 同样的对比了一下临床分期,结果与训练集一样
同样的对比了一下临床分期,结果与训练集一样
8、外部数据集验证
与上面同理
9、预后模型与LNR比较
LNR是一种新的、简单的标记物,可以很容易地对晚期胃癌的预后进行分层,我们使用我们构建的预后模型与LNR进行比较分析,看看他们之间有没有什么联系,
10、预后模型与TNM临床分期的关系
11、预后模型中的lncRNA的功能分析
- 发表于 2018-10-08 21:01
- 阅读 ( 7728 )
- 分类:文献解读
你可能感兴趣的文章
- 对方向扔了一个赤果果的生信分析SCI思路 9539 浏览
- miRNA与lncRNA的关系网络构建 13883 浏览
- 第三篇lncRNA signature的文章解析,IF五分胶质瘤 6233 浏览
- 两篇胃癌lncRNA signature文章解读之二(IF=3.2) 5806 浏览
- 人类lncRNA 简介 7228 浏览
